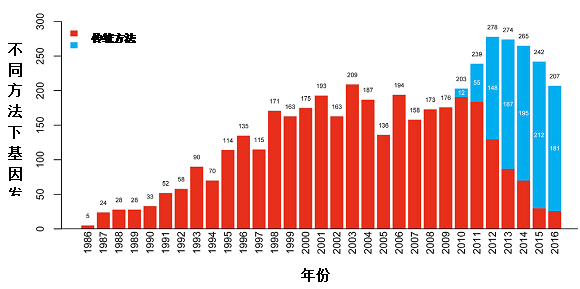
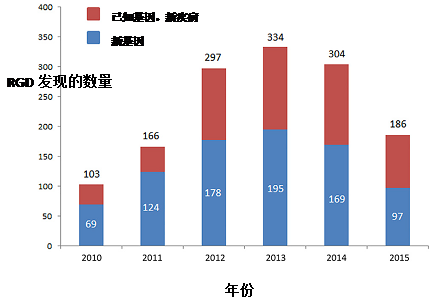
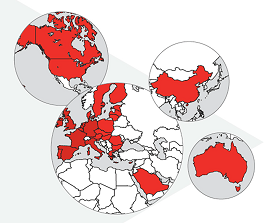
翻译:许璐、童流川
校对:许璐
本文由罕见病发展中心(CORD)编译,如需转载或引用请联系我们。
— 摘 要 — 为患有罕见遗传疾病的儿童和成人在分子层面及时进行确诊,可以缩短诊断时间、改善疾病管理,并有助于复发风险和生育选择方面的基因咨询。在目前,罕见病的临床确诊率约为50%;但对于在首次基因评估后没有获得分子诊断的患者,确诊率则大大降低。此类患者的诊断成功率很大程度上取决于罕见病相关基因及其致病机理的研究进度。因此,持续的科研活动对于构建更完整的疾病-基因关系是十分必要的。 2011年成立的国际罕见病研究联盟(International Rare Diseases Research Consortium,简称IRDiRC)旨在为罕见病相关科研人员和投资机构提供交流平台,实现对所有罕见病的分子诊断。本文综述了当今和未来基因探索过程中的瓶颈,并提出了改进方案。每一个成功的基因发现都将为相应的罕见病提供诊断、预防和治疗的机会,使精准医疗成为可能。 — 前 言 — 虽然罕见病中的每个单独类型都很罕见,但作为总体却很常见。罕见病在美国的定义为患病人数少于20万的疾病;1在欧洲的定义则为患病率低于1/2000。2许多罕见病是由于单个基因的功能改变所造成的,这些被称为罕见遗传病(Rare Genetic Disease,简称RGD),也被称为孟德尔遗传病或单基因病。 这些RGD影响了至少1/50的欧洲人口。3虽然我们对RGD数量的认识仍然有限,但根据现有的医学和基因证据推断,至少存在7000种不同的RGD4(参见“网络资源”中Orphanet的数据)。虽然RGD的进展一般是慢性的,但如果能及早诊断(例如进行新生儿筛查)和接受针对性的治疗,就可以缓解或推迟长期并发症的发生。 此外,确定性的分子诊断还有很多益处,如省去进一步的诊断、帮助获取合适的医疗资源、减少病程发展的不确定性、提供准确的复发风险咨询、促进正确的生育选择,以及向患者和家属传递社会心理学效益。重要的是,明确RGD背后的基因机制,将RGD与致病生物通路相联系,有助于对以往一些症状严重而且只能进行对症治疗(例如用ivacaftor治疗III型CFTR【MIM: 602421】病理性变异)5的疾病实现高效靶向治疗。精准医疗的最终实现将与RGD的成功诊断密不可分。 现阶段对RGD表型和基因多样性的了解 ———— ✤ ———— 虽然人们对RGD表型和基因多样性的了解正在逐步提高,但仍有明显的不足。确定RGD的具体数目因为种种原因而比较困难,其中包括区分新型RGD和已知RGD,将渐变的病理表现客观地区分为不同门类。目前有两个国际数据库为研究社群提供临床和基因数据:人类孟德尔遗传在线数据库(Online Mendelian Inheritance in Man,简称OMIM)4和Orphanet6。 OMIM自从1966年V. McKusick发表《人类的孟德尔遗传》以来就一直对孟德尔遗传病进行记录并分类,1987年上线并提供搜索服务。OMIM通过挖掘生物医学文献,结合专家评审,将基因和基因表型方面的新信息添加到相应的基因和表型目录;其关于孟德尔遗传病的各种数据被医学的各个分支学科在生物医学文献中广泛引用。OMIM通过将不同基因变异造成的相似表型分类到不同目录下,强调了基因和表型的关系;基因异质性则在相应的表型系列(Phenotypic Series)中得到体现。在最近的一项分析中(数据下载时间为2016年9月5日),OMIM已经收录了3209个基因和4550种相关的单基因罕见病。 Orphanet自1997年起进行遗传病和其它罕见病数据的收录和维护。在Orphanet数据库中,罕见病被定义一种可识别的、同质性的临床表现而无视其发病原因和相关基因的数目。Orphanet对罕见病进行多层级分类,并进一步分成多个亚型,包括基因亚型。Orphanet会进行文献检索,并收录新基因和新基因-疾病关系的相关文献,从而将数据库中的基因和疾病进行语义关联。截至2016年9月14日,Orphanet已经收录了3654个基因和3551种相关罕见病。 两个数据库中单基因遗传病数量的差异(OMIM 4550个,Orphanet 3551个)是由数据库构造的差别所导致的:OMIM将罕见病按照基因病源进行分类,而Orphanet则根据临床疾病鉴别进行分类,因此当同一种罕见病可由多种基因变异导致时,一个Orphanet条目可能包括多个OMIM条目。最近,临床基因组资源库(Clinical Genome Resource,ClinGen)7开始定义已发表的基因-疾病关联证据的强度。证据强度依照一个半量化的框架进行评分,最终评分和评分表以及数据来源一同发布在ClinGen网站。这些评分很快也将显示在OMIM数据库中。随着ClinGen的发展壮大,它将会更清晰地区分证据确凿的基因-疾病关系和需要更多证据的基因-疾病关系。 尽管罕见病基因背景的发现已经有了长足的进步,但仍有半数左右的罕见病尚未发现明确的病因。自80年代中期后的二十年以来,基因发现的手段主要是基于假设的连锁分析、定位克隆,以及候选基因或者区域基因的测序。自2009年起,“下一代”测序(next-generation sequencing,NGS)技术的引入加快了研究进度;它以全外显子组测序(whole-exome sequencing,WES)为基础,进行疾病相关基因的发现,且不需要提出假设。 如今,WES已被作为发现疾病-基因关系的首要手段进行常规应用。与全基因组测序(whole-genome sequencing,WGS)相比,WES的优势在于它显著的低成本,且绝大多数病理性变异都发生在基因组的蛋白质编码区域。毫无疑问,随着WGS成本的降低,临床医生和科研人员也将渐渐倾向于应用WGS技术,以利用其覆盖区域更广、能发现结构变异和非外显子区变异的优势。 对OMIM的分析发现,从2012到2015年(图1)每年平均有259种“新”RGD被发现,其中包括了157个疾病-新基因关系发现(此处定义为以往文献中没有与疾病进行关联的基因变异)和102个新疾病-基因关系发现(定义为以往文献报道中与其它疾病相关的基因变异;数据未展示)。8同期Orphanet平均每年新增281种RGD发现,包括160个疾病-新基因关系发现和121个新疾病-基因关系发现(图2)。 Orphanet和OMIM记录了几乎同样数量的疾病-新基因关系数量(平均分别为160个和157个),但Orphanet记录了更多的新疾病-基因关系发现(121个,OMIM为102个)。人工随机检阅发现,OMIM和Orphanet的区别可能主要在于收录过程的不同:OMIM更倾向于将文献报道认定为已知RGD症状的扩展,而不是新疾病-基因关系。即便如此,OMIM和Orphanet的数据都表明,RGD发现中有很大部分(分别为38% 和43%)属于由已知基因导致的新疾病(新疾病-基因关系)。这与最近一项对OMIM所有数据的分析相对照,呈现出一种有趣的趋势;后者发现,约有25%的孟德尔遗传病相关基因都与两种以上疾病相关联。 自WES技术兴起后,很多因严重影响生殖适应性而无法使用传统手段进行基因检测的RGD被发现与病理性从头突变(de novo pathogenic variants)或等位基因/位点的高异质性有关。这些RGD通常具有多种显著的临床表现,包括早年发病、具有严重的表型或者医学影像学特征,病因大多为高穿透性的病理性蛋白质编码区基因组变异。 此外,这些RGD一般属于常染色体遗传、X连锁隐性或从头突变显性,这些特性使它们更容易被WES技术所捕捉,也体现了WES基因发现体系的“最佳着力点”。OMIM和Orphanet的数据(图1和图2)都呈现出基因发现数量连年减少的趋势;这一趋势是否是真实并持续下去还需要未来几年的更多数据分析。可以确定的是,随着容易发现的RGD基因已被陆续检测出,想要保持甚至加快RGD的基因发现速度,目前存在的技术瓶颈亟待解决。 国际罕见病研究联盟 ———— ✤ ———— 国际罕见病研究联盟(International Rare Diseases Research Consortium,简称IRDiRC)成立于2011年,目的是为罕见病相关科研人员和投资机构提供沟通平台。IRDiRC会议由三个IRDiRC科学委员会(诊断、跨学科和治疗委员会)和来自三个患者倡导组织(包括来自美国的国家罕见病组织【National Organizationfor Rare Disorders,NORD】和基因联盟【Genetic Alliance】,以及来自欧洲的罕见病组织【Rare Diseases Europe-EURORDIS】)的代表共同主持(即曾经的执行委员会)。 会议成员包括公共研究资助机构和私人制药及生物技术研究所等共计42个成员组织,每个组织都承诺在其管辖权内至少投资一千万美元用于罕见病研究(图3;数据采样于2017年1月11日)。目前,经IRDiRC协调募集的罕见病研究资金已超过20亿美元。IRDiRC的目标是促进人们对所有罕见病的认识。 诊断和跨学科委员会(Diagnostics and Interdisciplinary Committees)以及相关组织的工作重点是发现当前和未来RGD探索的瓶颈,并提出可以通过国际合作解决的计划。我们预见到,想要保持甚至加快RGD研究的节奏,现阶段基因发现流程中的几个短板必须得到解决。这包括收集/分析临床和基因组数据、数据发掘和共享、致病原因发现的功能性支持,以及当前分析和基因组手段所检测不到的致病机理(表1)。 实现RGD全面诊断的策略 ———— ✤ ———— 未来数年内,对RGD背后分子机理的探究将对大型基础设施、资源和工具产生越来越多的需求。在过去的几年中,我们的委员会及相关组织划定了几个应当优先考虑的领域以帮助实现诊断全部RGD的目标。目前,IRDiRC提出一种名为“IRDiRC认证资源(IRDiRC Recognized Resources)”9的质量指标,该认证通过一些具体的标准凸显出核心资源(如平台,工具,标准,指南等)的重要性。如果加以广泛应用,可以加速RGD探索的步伐。 临床数据交换中的概念、术语和疾病分类 理解基因组的变化如何影响不同疾病表型对人类医学研究有着重要意义。如果没有仔细的表型特征记录,基因组数据即使再多,价值也必将受限。虽然我们在共享基因型数据方面有了很大进步,但在表型数据共享方面依旧缺乏具体的标准框架。对于无法确诊的RGD,由于世界范围内的相同病例寥寥无几,情况更加不容乐观。目前,多种概念、术语、分类的混杂使用,反映出不同群体在科研和医疗等多个领域所作出的努力,以及对标准框架的迫切需要。 IRDiRC认识到,表型标准用语、术语和疾病分类对RGD研究十分重要。人类表型标准用语联盟(Human Phenotype Ontology,HPO)10,11为RGD表型提供了有效的注释,并已被PhenomeCnetral、12DECIPHER13和UK10K计划14等RGD数据库所使用;应注意到,其它资源在特殊情况下也可能成为更合适的参考。 HPO系统已被一体化医学语言系统(United Medical Language System,UMLS)所整合,这使得更广泛的医学信息资源交换成为可能。HPO并不只是单纯的医学术语系统;它的词条按照层级分布,使计算机能够对不同诊断下的临床发现进行分析15,并可在进行WES分析之前对RGD表型进行临床16和基因发现17分层。标准用语发展的一个重点是提高HPO在罕见病冷门领域的精度和覆盖率。同时,建立纵向评估系统(评估发作及时间特性)、使用表型否定(患者不具有某种表型)以及记录量化指标(如实验数据的异常值)也是非常重要的。 为了提高不同系统之间的兼容性,解决特异性RGD术语缺乏的问题,新成立的国际人类表型术语联盟(InternationalConsortium for Human Phenotype Terminologies,ICHPT)为受众提供了常用表型术语的标准和定义以方便数据共享,特别是将RGD表型数据库和基因型数据库相关联。ICHPT由Orphanet(经由EuroGentest项目)、HPO18和OMIM(Robinson et al., 2014, Am. Soc.Hum. Genet., abstract)等组织共同创建,目前已提供超过2300项术语条目,并可由任一同义词进行检索。 这些条目已经被嵌入许多主流术语系统,包括HPO、11PhenoDB、19Orphanet、Elements of Morphology、20POSSUM、SNPMED、MeSH 以及MedDRA,促进了系统之间的兼容。虽然标准用语中包含许多更加具体和精细的条目,但这些条目都会被关联到更加广义的上级条目中。IRDiRC认可并鼓励将ICHPT作为分享表型数据时使用的最小标准用语集。 目前存在两个互补的罕见病疾病分类数据库:Orphanet罕见病分类(Orphanet Rare Disease Ontology,ORDO)21和OMIM。4ORDO是由Orphanet数据库衍生而成的结构化词汇库,它将疾病、基因和其他相关特征之间的关系进行汇总,形成一个利于计算机分析的资源库。ORDO整合了罕见病疾病分类、相关性(基因-疾病关系和流行病学数据)、其它术语库(MeSH、UMLS和MedDRA)、数据库(OMIM、UNIProtKB、HGNC、Ensembl、Reactome、IUPHAR和Geneatlas),以及各种分类(国际疾病和相关健康问题分类【ICD-10】)。 需要说明的是,ICD-10只有约500个罕见病分类代码。这一缺陷正在被Orphanet的多层级罕见病分类与编码(Orpha代码)系统所克服。这将成为ICD涵盖绝大多数已知罕见病的基础。Orpha代码正在逐渐被欧洲健康系统所采用,用于RGD信息的追踪记录。Orpha代码的引入也受到了国家罕见病计划与战略(National Action Plansand Strategies for Rare Diseases)项目的扶持,并被欧洲罕见病专家委员会所推荐。22 OMIM也在孟德尔遗传病的命名和分类中起了很大的作用。它对可识别特征进行了定义,并突出了可用于鉴别诊断的特征。一般而言,OMIM会根据基因病源的不同(即基因异质性)分别创建表型条目,每个表型的临床概要仅包括有文献报道的疾病相关基因变异引起的表型特征。每个OMIM表型都有唯一且确定的编号(MIM代码)用于上文提到的数据库和生物医学文献中。IRDiRC认可ORDO和OMIM的罕见病分类并支持二者间的持续合作。 促进基因组数据分析的标准、工具和资源 我们对基因组数据的分析、注释和共享能力是RGD研究进行的基础。目前,分析注释的工具和方法没有标准化,缺乏互通性;大规模基因组数据分析结果的共享因此受到了阻碍。DNA序列的分析流程还有很大的进步空间,包括序列比对、变异识别、功能注释和预测,尤其是在处理更复杂的包括插入、删除和广泛的结构变异时,需要统一的分析方法。23这一观点获得了近期数据的支持;数据显示,文献中报告的WES方法之所以收获有限(至少在某些隐性遗传病的背景下),主要是因为正确识别变异的能力不足。24 RD-Connect欧洲研究和诊断项目正在与EURenOmics和NeurOmics的RGD研究项目一起开发一个具有类似功能的平台。此外,还应当增强现有工具的互通性和普适性,并对它们的管理和更新进行妥善协调。 通过基因组数据分析进行RGD研究的另一项挑战是为罕见变异评定进一步解读的优先级。RGD的病因研究极度依赖于各种整合数据库所汇总的WES数据,如人类外显子组整合数据库(Exome AggregationConsortium,简称ExAC,包含60,000个外显子组)以及 NHLBI外显子组测序项目(Exome Sequencing Project ,简称ESP,包含6,500个外显子组)。这些数据库将不同疾病作为分析时的参考数据集,这一策略有效地将特定群体中的变异数目减少到了可以控制的数量范围。 然而,许多第一手的外显子比较数据集都来自西欧和北美人群,这限制了在样本数量较为稀少(或没有样本)的人群中的病理性变异的发现。千人基因组计划(1000 Genomes Project)作为一个大型异质性人群数据库,为增进我们对人类基因组的了解做出了突出贡献。最近,gnomAD已经积累了15,000个基因组和120,000外显子组数据,其中包括从千人基因组计划、ExAC和ESP获取的数据。增加此类人群相关数据集的数量,收集和分享RGD研究社群以及其他人类医学研究者需要、但在目前资料库中研究较少或缺失的人群数据,在未来将是十分重要的。 全球基因组学与健康联盟(Global Alliance for Genomics and Health,简称GA4GH)在这一领域非常活跃,它致力于通过联合生态系统方法(federated ecosystem approach)实现负责、高效的基因组和临床数据共享;我们支持GA4GH的做法及其在RGD领域的应用。25烽火网络(Beacon Network)是GA4GH的一个示范项目,它是一个全球化的搜索引擎,联结了来自各个人居大陆的60个基因变异数据库,实现了全球化的基因变异发现。 实践数据发现和共享的伦理标准 RGD研究社群对其在数据发现和共享方面的需求有着非常敏锐和普遍的认识.26鉴于我们面临的挑战是要了解并诊断更多且更罕见的RGD,最大限度地共享临床和基因数据已经成为至关重要的一环。在这方面,IRDiRC正与人类多样性组学项目(Human Variome Project,简称HVP)以及GA4GH展开合作,共同处理重大伦理、法律和社会问题,并建立了统一的国际数据标准以克服现存障碍。 IRDiRC认可《基因组学与健康相关数据负责任的共享框架》(Framework for Responsible Sharing of Genomic and Health Related Data)的主张27,该框架以在国际上遵循《世界人权宣言》(UN Declaration of Human Rights)第二十七条为前提,后者主张人人享有“共享科学进步及其惠益”的权利,以及“保护作者自科学研究成果中获得的精神及物质利益”。最近,针对《数据转移协议》的建议和模型已经发表,并被标注为“IRDiRC认证资源”。29 IRDiRC、HVP以及GA4GH之间的协作正在为制订国际公认的数据共享标准铺平道路。数据共享管理的几个关键领域是目前合作的重点。首先,合作制订了“分层”许可的政策,后者取决于数据收集和使用(临床或科研)的背景以及共享数据被识别的风险级别;该政策目前正在被MME数据交换平台(Matchmaker Exchange;见下文)30,31使用。 两个相关计划,即知情条款(Consent Codes)32模型以及自动发现和访问矩阵(Automatable Discovery and AccessMatrix,ADA-M),正在尝试对科研及临床记录相关内容的知情、合法、制度化的许可和限制进行系统化陈述,以促进流线型的数据发现、共享和使用。这也有助于更好地规范知情同意书条款,从而对科研和伦理审查委员会的作业进行指导优化。正如知情同意的操作需要增强交互性以实现更大规模的数据共享,数据访问机制也是如此。目前一种新的模型正在开发中,它将会方便数据访问(注册访问)并通过对用户进行标准化在线授权认证实现与MME等项目的交互。 注册访问将对不同类型的潜在数据用户(研究人员、临床护理专业人员以及患者)进行区分,并将数据按照可辨识度(identifiability)和敏感度(sensitivity)进行分级。IRDiRC与GA4GH之间正开展更多合作,以开发一种能够关联同一患者在多个项目中的不同数据并同时尊重个人隐私的隐私保护联动系统。促进国际项目和协会伦理审查精简化和统一化的伦理审查政策也已出台。随着时间的推移,以上种种努力将使本地的伦理、法律、社会政策和程序协调一致,以实现高效、负责的基因组数据及临床数据的国际化共享和分析。 支持基因发现的基因证据 来自包括加拿大FORGE联盟(FORGE Canada Consortium)33、美国孟德尔基因组学中心(US Centers for Mendelian Genomics)8和英国发育障碍解密研究会(UK Deciphering of Developmental Disorders study)34在内的几个大规模合作研究项目的报告显示,在非常特定的条件下(包括针对多个具有相同症状的家庭进行系统的表型探查),RGD的“解析率”(solve rate)通常大于50%。在不同临床诊断环境下、超过9000例患者的致病基因变异研究中,诊断的总体成功率则为30%左右。35–39这些近期的队列研究发现,成功临床诊断中的很大一部分(25%–30%)有赖于疾病相关基因的最新研究进展。科研背景下更高的解析率表明,在未解决的临床队列当中仍隐藏着许多可能的发现。 以病例为依据的基因发现匹配 根据以往的经验,在一名患者或一个家系中检测到的基因变异结果发表后,在另一名无亲缘关系的患者身上发现类似变异所间隔的时间约为2-3年。因此,高效地鉴定出更多在相同基因位点存在病理性变异并具有相似表型的无亲缘关系患者是一项核心挑战。一些漏网的候选基因(包括在隔离数据、通路以及模式生物文献中层层筛查仍未发现的有害变异)尚未见于文献报道或处于当前无法研究的“孤立”状态,测量它们的数量十分困难,但估计应超过1000种。 为应对这一挑战,许多合作项目开发了基于基因和表型的匹配算法12,13,40–52;然而,这些项目之间仍然缺乏沟通。就在前不久,IRDiRC诊断科学委员会(IRDiRC Diagnostics Scientific Committee)与Can-SHARE和GA4GH等数据分享服务的参与者合作推出了名为MME的联合数据平台。53该平台通过标准化的应用程序界面(application programming interface,API)和标准化的操作流程易化了针对具有相似表型和基因型的患者和家庭的基因鉴定。40 MME使得对多个数据库的同步搜索成为可能,避免了向不同检索服务多次提交数据的麻烦。在初始API界面下,每个服务器可以自行调整参数:匹配达成所要求的相似度(可以是基因型或表型)由不同的数据服务项目自行设置。MME平台的上线是非常重要的一步,目前来自PhenomeCentral12、GeneMatcher41、DECIPHER13、MyGene2 54、matchbox和Patient Archive的超过20,000例无亲缘RGD患者的数据已经实现了共享。然而,真正实现基于病例的基因匹配最优化、RGD基因发现的全球化,还需要国际数据共享的改进优化以及财政支持,并扩大基础设施、操作流程和算法的规模。 支持基因发现的功能证据 基因组数据向系统生物学的整合 在通过WES进行基因分析取得巨大进展的同时,其它有助于RGD发现和确认未知变异的大规模组学(如蛋白质组学、转录组学和代谢组学)项目也在开展中。例如,当WES发现了多个可能的基因后,蛋白质表达水平和功能的变化有助于确定是哪一种基因变异导致了疾病的发生。不同组学数据库在患者群体或个体水平上的数据整合有助于在高表型多样性(high phenotypic variability)和不完全外显(incomplete penetrance)的情况下了解疾病相关基因变异的重要性,以及帮助诊断和治疗用生物标记的研发,在靶向治疗的研究也将扮演重要角色。此类项目的数量和可持续性都有待增加。 促进基因发现的模式系统 对于模式系统(Model-systems),如人类、酵母、果蝇、线虫、斑马鱼、小鼠等生物的研究,对阐明候选基因的变异在疾病当中的作用、发现和确认新的药物靶点以及其它治疗策略都有重要的作用。发现新基因位点的速度已经超过了我们理解基因、通路和功能网络中各种突变的生物学效应的速度。有一种方法可以在新一代疾病模型中以高效、经济、通用并且高通量的方式弥补这一裂隙。这需要更完善的基础设施以实现下列目标: (1)使发现致病基因变异临床医生能够接触任何现有的实验工具; (2)允许研究各种模式生物的专家应用他们的技术解决相关的生物和临床问题; (3)提高效率,避免重复的研究,并使现有模型的利用率最大化。这些努力的最终目标是及早实现临床医生和科研工作者之间的沟通并为合作项目提供种子基金。 加速临床医生和科研工作者之间合作的一种策略是主动寻求“对口”的合作项目,并提供项目启动的种子基金。加拿大正在进行一项国家级的基础设施建设,这种名为“罕见病:模型与机制”(Rare Diseases: Models and Mechanisms)的网络会在发现疾病相关基因的第一时间将临床医生和科研工作者进行联系。56 该计划正处于三年资助周期的第二年,它已经促成了40多名医生和科研工作者之间的合作匹配。另一种办法是“助力”策略,即国家基金审批部门允许研究者们合作申请对已有项目的额外资助。在美国,对“R”类项目和“P”类项目的行政补贴并不鲜见;事实上,这也是NIH未确诊疾病项目(NIH Undiagnosed Disease Program)针对已经发现候选基因的研究项目的资助策略。57帮助临床医生寻找相关课题研究者的综合性国际虚拟网络也是一种补充和过渡策略。 促进与其它生物相关联的新型表型鉴定流程的建立和验证也很重要,其中的要点包括疾病相关性、病生理通路、候选基因,以及高效率。这也将促进对基因组变异和候选基因的评估、检测疾病相关指标的药物及靶点测试,并加深对疾病机制和病理过程的理解。在某些情况下,表型鉴定流程能够评估与人类疾病显著相似的特征(例如畸形和病理性行为特征等)。 如果表型足够特异(即为某种疾病所特有),则可直接确认其与疾病模型的相关性。自2009年以来,Monarch Initiative一直致力于这一领域的工作,它作为数据整合与分析平台,将不同物种的表型和基因型相互联系。同时,表型鉴定流程也能对直系同源表型(orthologous phenotypes,又名‘‘phenologs”)进行评估。 这类表型看似与目标疾病没有显著关联,却是由相同的分子水平缺陷导致的。58此外,找出新型、可靠、高效、疾病相关检测的模式表型和能够进行跨物种交叉比对的表型(即平行表型鉴定,parallel phenotyping)也同样重要。确认后的跨物种疾病相关表型将为克服现阶段瓶颈提供所需的检测结果,例如大规模确认等位基因和疾病相关基因。这正是后基因组测序时代所迫切需要的。 新的疾病机制 ———— ✤ ———— RGD基因研究在过去的几年里取得了巨大的进展。然而,尽管许多研究小组使用WES(有时是WGS)方法进行了不少研究,依旧有不少著名罕见病(如Hallerman-Streiff 综合征、Dubowitz综合征、VACTERL、Gomez-Lopez-Hernandez综合征、Aicardi综合征和PHACE综合征等)的遗传机制尚不清楚。研究失败的原因多种多样,最可能的原因一是技术方面的限制(例如注释错误、编码和非编码区域变异以及结构变异的遗漏),二是复杂的生物学特性(例如极端的位点异质性、组织特异的体细胞嵌合、罕见的遗传模式、家系间等位基因或基因位点异质性,以及致病的同义变异)。 克服以上障碍的方法并不多。不仅如此,罕见基因病如果不能通过现有的WES方法简单地找到答案,阐明其基因机制将会难上加难。想要为当前研究手段下感到棘手的疾病找到分子基础,需要在基因发现策略的应用方面更加广泛和创新(例如WGS、受累细胞和组织的RNA测序,以及对三种主要胚胎细胞谱系来源的组织的深度测序);改进计算和统计模型,优化变异鉴定、注释、功能预测和优先级评定——尤其是对于非编码区的基因变异;59还应当开发新的致病机理研究策略。 此外,一些项目以医生和专家的丰富经验和围绕患者个体进行的精确表型鉴定为优势,把跨学科评估作为目前努力的重点,比如国际未确诊疾病网络(Undiagnosed Diseases Network International)。60以上策略的开展和应用将进一步刺激对通过基因和功能途径阐明致病机理的资金支持。 关键的后续步骤 ———— ✤ ———— IRDiRC的目标是通过一种方法诊断所有的RGD,达成这一目标需要了解所有疾病的基因机制。这是一项具有重要意义的挑战,它正催生出一套包含所有RGD和相应基因变异的完整表型特征目录,引导人们寻找在非经典遗传模式下鉴定RGD病因的方法,创造用新知识治疗患者的工具和资源(例如,对基于NGS方法的国际临床应用指南进行统一和采纳)。 要完成这项挑战只能依靠重大国际合作和利益相关者们大规模的参与。IRDiRC和GA4GH等让科研社群加入的尝试具有重要的意义,也需要国际性的协调和资助。改进对罕见病患者的基因组临床分析中的翻译和报销策略是必要的;这对于避免在基因发现项目中重复鉴定大量已知基因的病理性变异,并将科研资金重新分配到新基因的发现和验证中尤为重要。让临床实验室、研究者和患者群体共同参与到数据共享中也十分关键。 我们还必须认识到,随着更多基因与人类疾病的相关性被发现以及适当分析方法的确立,RGD诊断所面临的一项重要挑战始终存在:对数量不断增长的意义不明变异进行解析。RGD的DNA诊断主要依靠基因、基因组变异和表型方面的知识共享。目前,诊断数据由许多不同的诊断实验室通过丰富的手段进行收集并储存在大量不同的服务器系统和数据库中,它们之间大多缺乏联系,处于“筒仓式”存储状态。 因此,需要给出本地解决方案,以实现简便、可重复、使用通用标准和术语的基因变异及相关表型的数据存储。此外,这些本地系统还需要实现全球化联结以构成一个“基因知识网络”。对患者正常治疗标准的共享需要群体性参与,因此有必要整合现有平台(例如ClinVar61、Leiden Open Variation Database【LOVD】62和DECIPHER13)上的临床基因和表型数据,将不同类型的数据(如矩阵和测序)相互联系,并涵盖从小型(单核苷酸)到大型(缺失、重复、倒位等)变异。 数据库中存在的致病基因判定假阳性会使这项工作进一步复杂化,因此由相关专家进行的数据管理将最终成为提高诊断精确度的关键。与RGD发现一样,基因变异的病理性/良性分类也高度依赖于多种工具和方法,特别是不同范围的人群特异的疾病和控制数据库、利用正交分析如代谢组学、转录组学、蛋白组学等阐明功能,以及在易操作的模式生物和细胞中进行疾病相关基因变异的系统性扫描。显然,为每个基因变异找到病因是信息指导下的患者治疗的关键。 找到能够诊断所有RGD的方法对患者及其家庭意义重大。它将实现基因咨询、更准确的预测、针对个体鉴定特定健康风险,以及避免不必要或有害的诊断性介入和治疗。最终,这些知识将被应用于全基因组范围的测序,对新生儿进行诊断和筛查。64面对不断增长的患者人群,只有在诊断结果确定的情况下(如溶酶体贮积症、先天性肌无力综合征)才能进行有效的药物治疗。65 这一目标也使更多患者能够参与到要求绝对的分子和表型诊断证据的临床研究队列当中,为学术机构和私人研究所正在研发的新药和介入手段提供潜在的益处。66在我们看来,对所有RGD的了解将成为实现精准医疗的转折点;用基因组的力量对罕见病进行解释,伴随对生物学过程的深刻理解,将为患者和他们的家庭带来医疗技术的迅速转变。 图1. OMIM数据库2010年以来基于WES和WGS的基因发现数量与传统方法的对比 自WES和WGS技术2010年问世以来,RGD基因发现的步伐逐年加快,WES和WGS方法下发现基因的数目(蓝色)相对传统方法(红色)比重逐年增加。2013年之后,WES和WGS发现基因的数目几乎是传统方法下的三倍,但基因发现速度逐渐变慢。(图表修改自Chong etal.8) 图2. Ophanet数据库2010-2015年的新基因-表型发现统计 2010年以来,基因-新表型关系(已知基因与新疾病的联系)的发现比例逐年增加。2013年以来,新基因和基因-新表型关系的发现速度逐渐减慢。 图3. IRDiRC成员分布图 IRDiRC成立于2011年,目前拥有来自亚洲、中东、澳洲、欧洲及北美的42个成员机构。来自上述公共及私人机构的罕见病项目投资总计已达20亿美元。 表1基因发现流程中的瓶颈因素 临床数据 非特异的临床表现(例如发育迟缓和肌张力低下) 高度稀有的和未发现的基因病 包含完整人类表型谱系的标准用语的缺乏 标准用语或3D面部特征分析在表型鉴定当中的不完全应用 跨学科患者评估方法的不一致性 无法对年龄特异的疾病特征进行解释和比较 基因组数据 WES的技术限制(例如无法准确识别拷贝数量变异和结构变异) 标准化技术和信息方法的缺乏 不完整的人群特异对照数据集 数据发现与共享 缺乏广泛应用的数据共享框架 缺乏通用的数据共享标准 缺乏系统性记录数据使用条件的方法 缺乏针对每名研究参与者的隐私保护联动系统 基因证据 孤立的数据集 数据共享基础设施的缺乏和使用 功能证据 缺乏标准化和中通量的变异影响分析 缺乏对大多数人类基因功能的生物学知识 新的疾病机制 缺乏非编码基因变异分析的专业知识 其它机制,包括组织特异性嵌合、甲基化、寡/双基因遗传 网络资源 1000Genomes, http://www.1000genomes. Can-SHARE,http://www.p3g.org/resources/can-share ClinGen, https://www.clinicalgenome.org ClinVar, http://www.ncbi.nlm.nih.gov/clinvar DECIPHER, https://decipher.sanger.ac.uk EURenOmics,http://eurenomics.eu ExomeAggregation Consortium (ExAC) Browser, http://exac.broadinstitute.org GeneMatcher,https://genematcher.org GenomicsEngland, https://www.genomicsengland.co.uk Genotypeto Mendelian Phenotype (Geno2MP), http://geno2mp.gs.washington.edu GlobalAlliance for Genomics and Health(GA4GH), http://genomicsandhealth.org gnomAD, http://gnomAD.broadinstitute.org HumanPhenotype Ontology (HPO), http://www.human-phenotype-ontology.org HumanVariome Project, http://www.humanvariomeproject.org/ InternationalConsortium of HumanPhenotype Ontologies (ICHPT), http://www.irdirc.org/ichpt InternationalRare Diseases Research Consortium(IRDiRC), http://www.irdirc.org LeidenOpen Variation Database (LOVD),http://www.lovd.nl/3.0/home Matchbox, https://seqr.broadinstitute.org MatchmakerExchange (MME), http://www.matchmakerexchange.org MedicalSubject Headings (MeSH), http://www.ncbi.nlm.nih.gov/mesh MedicalDictionary for Regulatory Activities(MedDRA), https://www.meddra.org MonarchInitiative, https://monarchinitiative.org MyGene2, http://mygene2.org NeurOmics,http://rd-neuromics.eu NHLBIExome Sequencing Project (ESP)Exome Variant Server, http://evs.gs.washington.edu/EVS OMIM, http://omim.org Orphanet, http://www.orpha.net OrphanetRare Disease Ontology, http://bioportal.bioontology.org/ontologies/ORDO OrphanetRD-Action, http://www.rdaction.eu PatientArchive, http://patientarchive.org PhenoDB, https://phenodb.org PhenomeCentral,https://www.phenomecentral.org POSSUM, http://www.possum.net.au RareDiseases Models and MechanismsNetwork (RDMM), http://rare-diseasescatalyst-network.ca RD-Connect,https://platform.rd-connect.eu SNOMED CT,http://www.ihtsdo.org/snomed-ct UK10K, http://www.uk10k.org 参考文献 (下略)